
Convex Hull
Why Convex Hull?
이전 포스팅 ( T-SNE IN R/PYTHON )에 이어서 t-SNE를 사용한 맵핑 결과를 그래프에 그려보려고 한다. 지금 예제에서 사용한 Iris 데이터셋은 150 row라서 산점도로 그려도 크게 무리가 없다. 하지만 데이터가 엄청나게 많아진다면? 그룹 수도 많아지고 1000만 row를 넘어가는 결과물에 대한 클러스터링이라면 어떻게 그려야 효과적으로 전달할 수 있을까?
일단 1000만 row 정도 되는 경우 scatter plot을 그리는 것은 전달 방식을 떠나서 그래프를 그리는 것부터 문제가 된다. (특히나 svg로 그릴 경우 비효율적이다. 겹쳐서 보이지도 않는 점들을 모두 렌더링 해야하기 때문에) 웹으로 시각화 결과물을 전달해야 하는 경우라고 보면, DOM과 svg를 적극 활용하는 D3에서 이런 결과물을 렌더링하는 순간 위이이잉 하는 비행기 이륙소리가 들릴 것이다. WebGL까지 사용한다면 클라이언트의 리소스를 최대한 활용하여 보여줄 수는 있겠지만, 불필요한 정보량을 줄여서 간단하게 보여줄 수는 없을까??
정보를 가득가득 채워서 2-D Density 를 구하고 contour 형태로 보여주는 것도 한 가지 방법이겠다. 하지만 밀도까지는 필요없고 그냥 해당 클러스터가 차지하는 영역을 알고자 하는 정도라면, Convex Hull을 구해서 빠르고 간단하게 클러스터를 그려볼 수 있다.
Convex Hull
Convex Hull에 대한 수학적인 정의는 위키피디아 를 살펴보는 것으로 하자. 단순하게 정리해보면 평면 위에 존재하는 집합 X 전체를 감싸기 위해 필요한 최소한의 부분 집합이라고 생각하면 될 것 같다. 수학적으로 엄밀한 설명은 아니겠지만, 그래프를 그리기 위한 설명으로는 각 클러스터에서 가장 바깥에 있는 점을 의미한다. 따라서 가장 바깥에 있는 점들만 서로 이어그리면, 그 안쪽의 공간은 클러스터가 차지하는 공간이 된다.
Convex Hull을 이용한 클러스터링 시각화
이제 R과 파이썬을 사용해서 직접 Convex Hull을 찾고 그려보자. 저번 포스팅에서 이어지기 때문에, 앞 부분에 대한 설명은 생략한다.
R
library('tidyverse')
set.seed(1)
iris_tsne = tsne::tsne(as.matrix(iris[1:4]))
df_iris_tsne = iris_tsne %>%
as.data.frame() %>%
tbl_df() %>%
mutate(species = iris$Species)
result_dbscan = fpc::dbscan(iris_tsne, eps = 3)
R에서는 내장된 chull 함수를 이용하면 convex hull을 구할 수 있다. chull 함수를 통해 convex hull을 구할 경우 몇 번째 점들이 가장 바깥에 존재하는지 index에 해당하는 벡터를 반환한다. 따라서 원래의 데이터프레임을 chull 함수로 구한 값과 매칭되는 row로만 필터링하면 우리가 원하는 결과물 (Convex Hull에 해당되는, 가장 바깥 포인트들의 x,y좌표) 을 얻을 수 있다.
아래 코드에서는 클러스터별로 convex hull을 구하기 위해 dplyr::group_by 와 dplyr::do를 사용해서, 클러스터별로 convex hull을 구한다. 그리고 dplyr::slice 함수를 통해 우리가 구한 row에 해당하는 값만 추출한다.
df_chull_by_cluster = df_iris_tsne %>%
mutate(cluster = factor(result_dbscan$cluster)) %>%
group_by(cluster) %>%
do(slice(., chull(.$V1, .$V2))) %>%
ungroup %>%
select(V1, V2, cluster)
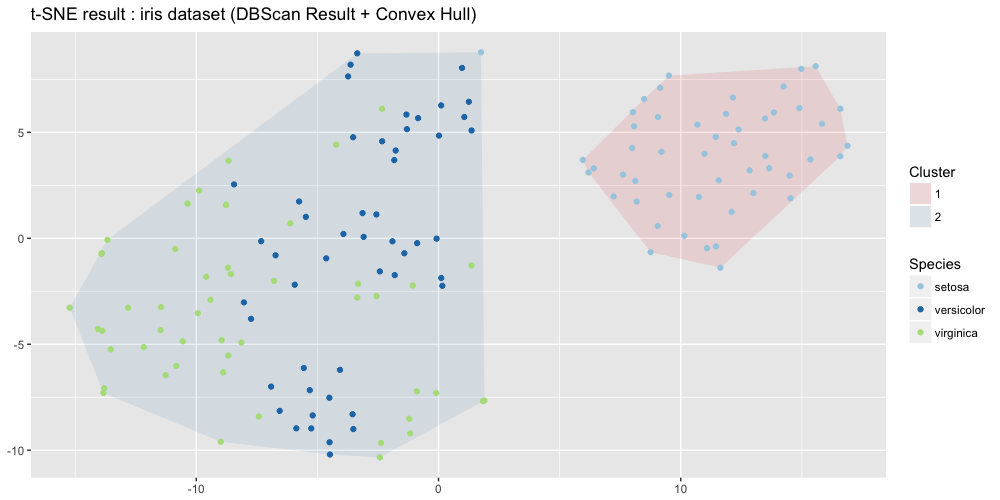
이제 convex hull에 해당하는 포인트를 구했으니 그래프에 표시해보자. ggplot2::geom_polygon을 통해 해당 구역의 면적을 표시할 수 있다. 투명도를 0.1 정도로 낮게 주고, geom_point를 뒤에 두어서 클러스터의 면적은 은은하게 바닥에 깔려있도록 하는 것이 좋다. (점 위에 면을 깔아버리면 색상에 따라서는 점이 잘 안보이고 완전 묻혀버릴 수도 있다)
# 짜잔
df_iris_tsne %>%
ggplot(aes(x = V1, y = V2)) +
geom_polygon(data = df_chull_by_cluster, aes(fill = cluster), alpha = 0.1) +
geom_point(aes(color = factor(species))) +
scale_color_brewer(name = 'Species', palette = 'Paired') +
scale_fill_brewer(name = 'Cluster', palette = 'Set1') +
ggtitle('t-SNE result : iris dataset (DBScan Result + Convex Hull)') +
theme(axis.title.x = element_blank(),
axis.title.y = element_blank())
Python
이번에도 역시 앞부분은 저번 포스팅과 동일한 내용이다. 다만 그래프 라이브러리를 altair에서 plotnine으로 변경했다. (ggplot2와 거의 동일하게 사용할 수 있어서 마음이 편안해진다…) plotnine 에 대한 자세한 설명은 https://plotnine.readthedocs.io/ 에서 확인해보자.
import pandas as pd
import numpy as np
from sklearn.manifold import TSNE
from sklearn.cluster import DBSCAN
from scipy.spatial import ConvexHull
from plotnine import *
%matplotlib inline
%config InlineBackend.figure_format = 'retina'
iris = pd.read_csv('https://raw.githubusercontent.com/mwaskom/seaborn-data/master/iris.csv')
np.random.seed(1)
iris_matrix = iris.iloc[:, 0:4].values
iris_tsne_result = TSNE(learning_rate=300, init='pca').fit_transform(iris_matrix)
df_iris_tsne_result = (
pd.DataFrame(iris_tsne_result, columns=['V1', 'V2'])
.assign(species = iris['species'])
)
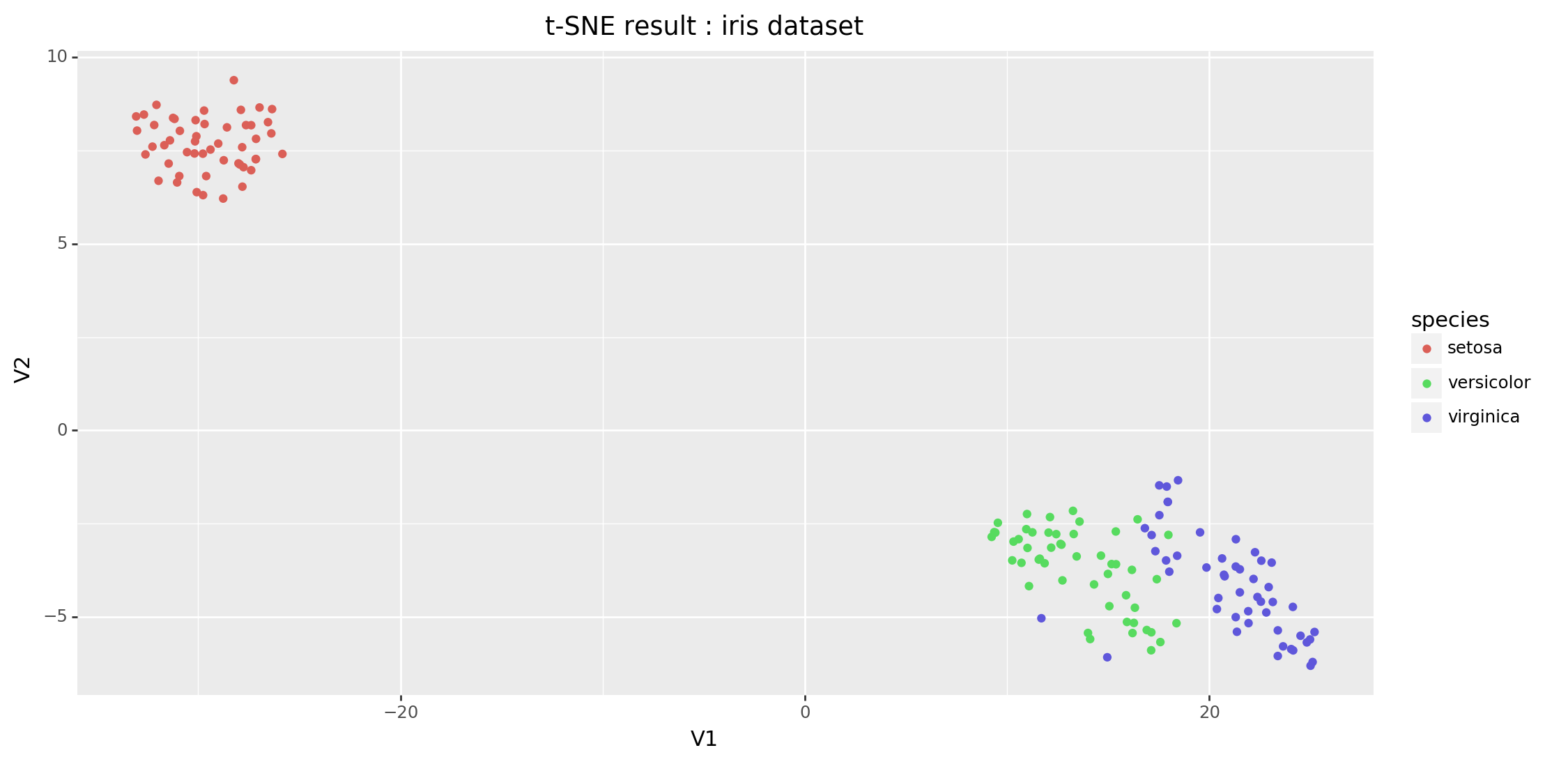
plotnine 으로 t-SNE 맵핑 결과는 아래와 같이 그릴 수 있다. 라이브러리를 업데이트 했더니 seed가 바뀐건지 저번 포스팅과 결과물이 달라졌다…..ㅠㅠ
(ggplot(df_iris_tsne_result, aes('V1', 'V2', color='species')) +
geom_point() +
ggtitle('t-SNE result : iris dataset') +
theme(figure_size=(12, 6))
)
DBScan은 scikit-learn의 sklearn.cluster.DBSCAN을 사용하기로 했다. .fit_predict() 메서드를 사용하면 각 포인트별로 어떤 클러스터에 해당되는지 반환된다.
iris_dbscan_result = DBSCAN(eps=5).fit_predict(iris_tsne_result)
df_iris_dbscan_result = (df_iris_tsne_result
.assign(cluster = iris_dbscan_result)
)
Convex Hull은 scipy.spatial.ConvexHull을 사용한다. pandas DataFrame에서 클러스터별로 한꺼번에 처리하기 위해서 group_chull 이라는 함수를 만들고, groupby 와 apply를 통해 각 클러스터의 convex hull을 계산했다.
def group_chull(df):
ch = ConvexHull(df.loc[:, ['V1', 'V2']].values)
return df.iloc[ch.vertices, :]
df_chull_result = (df_iris_dbscan_result
.groupby('cluster', as_index=False, group_keys=False)
.apply(group_chull)
.reset_index(drop=True)
.loc[:, ['V1', 'V2', 'cluster']]
)
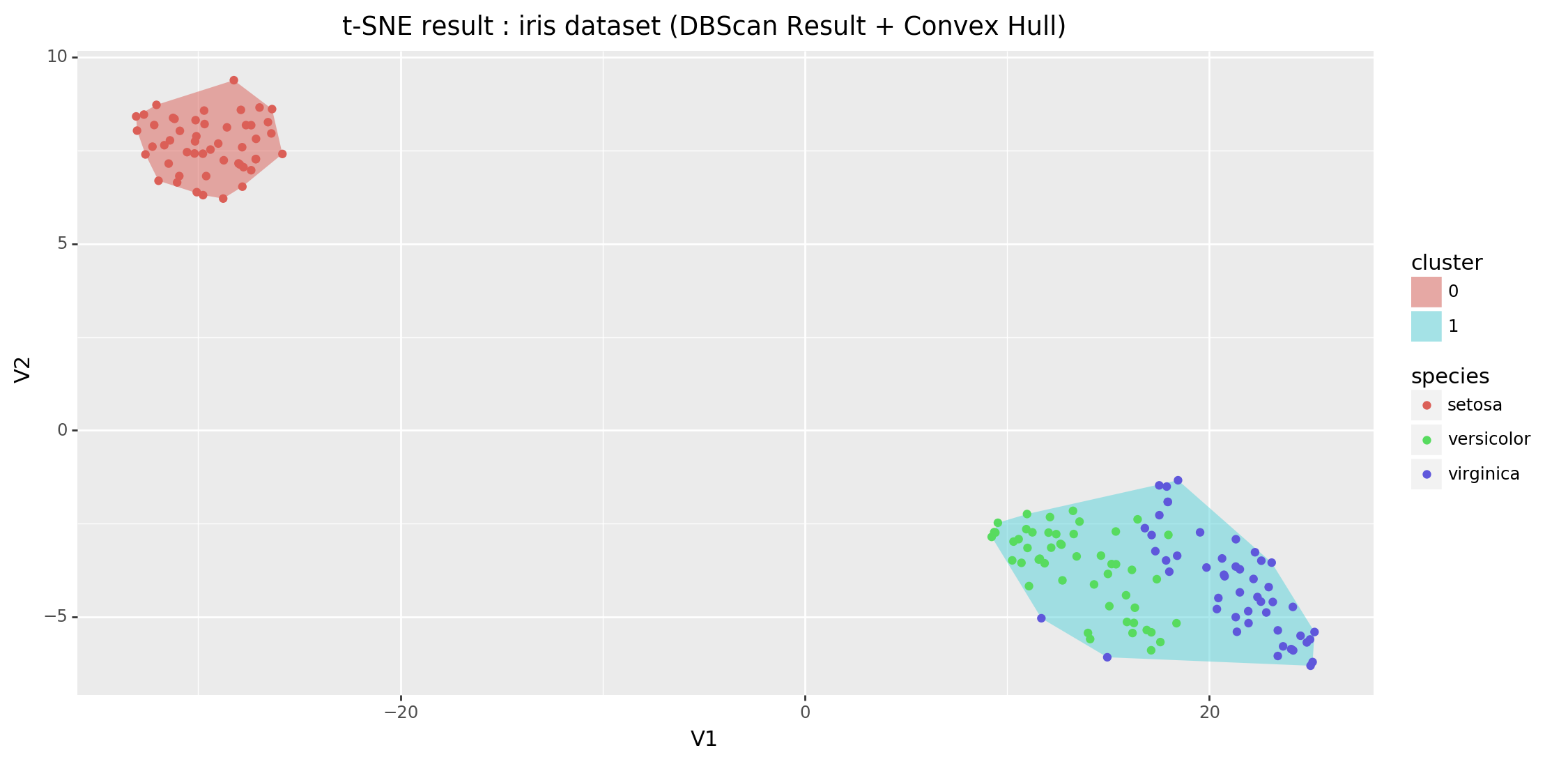
이제 결과물을 그래프에 그려보자. 여기서 주의할 점은 현재 cluster 변수가 int 타입으로 되어 있다는 것이다. 따라서 그냥 색상을 적용하게 되면 nominal / categorical 팔레트가 아니라 Sequential 팔레트가 적용된다. 따라서 여기서는 cluster 변수를 문자열로 바꿔두고 (.astype) 그래프를 그린다.
(df_chull_result
.assign(cluster = lambda d: d['cluster'].astype('str') )
.pipe(ggplot, aes(x='V1', y='V2')) +
geom_polygon(aes(fill='cluster'), alpha=0.5) +
geom_point(data=df_iris_dbscan_result.assign(cluster = lambda d: d['cluster'].astype('str')),
mapping=aes(color='species')) +
ggtitle('t-SNE result : iris dataset (DBScan Result + Convex Hull)') +
theme(figure_size=(12,6))
)
마무리
분석을 하는 과정에서 그래프를 그릴 일이 정말 많이 생긴다. 그런데 분석과 인사이트를 위해 혼자서 그래프를 그려볼 때와, 누군가에게 하고자 하는 말이 있어서 정리를 할 때는 그리는 방식이 달라지는 것 같다. 분석을 위해 그릴 때는 그래프 안에 정보량을 많이 담아서 놓치는 것이 없도록 하는 경우가 많다. 하지만 어느 정도 하고자 하는 말이 정해질 경우 불필요한 요소는 점점 정리하고 정말 필요한 요소만 남기게 된다.
예를 들어 사용자들의 구매를 기준으로 상품을 2차원 공간에 맵핑해서 클러스터링을 한다고 해보자. 서비스에서 판매하는 상품이 몇 천개를 넘는다면 모든 상품을 다 점으로 찍을 수는 없는 일이다. 이럴 때는 말하고자 하는 바에 의해서 그리는 방식이 결정된다. 잘 팔리는 상품 100개에 대한 이야기를 한다면 상위 100개 상품을 뽑아서 그리면 된다. 추출한 클러스터가 명확한 특징을 가지고 있어서 그에 대한 이야기를 한다면 위와 같이 convex hull을 구해서 적당히 면적 표시하고 각 클러스터에 적당한 레이블만 달아서 설명을 할 수도 있다.
위에서 설명한 클러스터링 시각화 같은 것들은 사실 모른다고 해서 분석이 안된다거나 하는 것은 아니다. 다만 적절하게 활용할 수 있다면 작은 디테일 하나를 얹을 수 있게 된다. 그리고 가끔은 이런 디테일의 유무로 많은 것들이 바뀌기도 하니깐 뭐.

