
Preprocessing the data
데이터를 분석하다 보면 분석하기 전에 전처리를 수행해야 하는 경우가 많이 발생한다. 전처리를 위해 직접 코드를 짜는 경우도 있고, 다른 패키지에서 제공하는 기능을 사용할 수 있다. 여기서는 caret 패키지의 preProcess 함수를 기준으로 해서 함수를 적용하는 방법에 대해 설명하려고 한다.
Feature transformations
caret 패키지에서는 preProcess 함수를 통해서 표준화 등의 전처리를 수행할 수 있다. caret을 이용한 변수변환은 두 개의 단계로 구성된다.
우선 preProcess 함수를 사용해서 변수변환에 필요한 parameter를 설정한다
그리고 predict 함수를 통해 실제로 값을 변화시킨다
R에서 모델을 구성하고 예측하는 방법과 동일한 구조를 취하고 있기 때문에 기존에 R을 사용하던 사람이라면 쉽게 익숙해질 수 있다.
preProcess 함수의 첫 번째 인자는 변환시키고자 하는 데이터이다. data.frame의 형태를 가지는 수치 데이터를 요구한다
두 번째 인자는 변환 방식이다. "range", "BoxCox", c("center", "scale"), "pca" 등 다양한 방식을 제공한다. 기본값은 c("center", "scale")이다
Z-Score normalization
먼저 살펴볼 변수 변환 방식은 z-score를 이용한 정규화이다. 데이터가 정규분포에 가까운 형태를 가질 경우에 생각해 볼 수 있는 방법이다. 각 변수들이 종모양의 형태를 보이지만 단위에서 큰 차이를 보일 때, 분포는 동일하게 유지하면서 값의 단위를 비슷한 수준으로 맞출 수 있다
preProcess함수를 적용하면 변환에 대한 정보를 살펴볼 수 있다. 수치형 자료가 아닌 Species 열은 무시된다
# install.packages('caret')
# install.packages('e1071')
library(caret)
zs_iris = preProcess(iris, method = c("center", "scale"))
zs_iris
## Created from 150 samples and 5 variables
##
## Pre-processing:
## - centered (4)
## - ignored (1)
## - scaled (4)
method = "center"는 데이터에서 평균값을 뺀다
method = "scale"은 데이터를 표준편차로 나눈다
따라서 두 옵션을 모두 사용하면 Z-Score를 사용한 정규화를 시행할 수 있다. 변환시킨 변수는 평균이 0이고 분산이 1이 된다
zs_iris_t = predict(zs_iris, iris)
head(zs_iris_t)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 -0.8976739 1.01560199 -1.335752 -1.311052 setosa
## 2 -1.1392005 -0.13153881 -1.335752 -1.311052 setosa
## 3 -1.3807271 0.32731751 -1.392399 -1.311052 setosa
## 4 -1.5014904 0.09788935 -1.279104 -1.311052 setosa
## 5 -1.0184372 1.24503015 -1.335752 -1.311052 setosa
## 6 -0.5353840 1.93331463 -1.165809 -1.048667 setosa
mean(zs_iris_t$Sepal.Length)
## [1] -4.484318e-16
var(zs_iris_t$Sepal.Length)
## [1] 1
Feature Scaling (Unity-based normalization)
데이터가 균일하게 분포되어있는 경우에는 [0, 1] 범위로 값을 조정하는 것이 도움이 될 수 있다. 데이터의 최대값을 1로 두고 최소값을 0으로 할 때, 각 데이터의 상대적인 위치를 값으로 한다.
un_iris = preProcess(iris, method = "range")
un_iris
## Created from 150 samples and 5 variables
##
## Pre-processing:
## - ignored (1)
## - re-scaling to [0, 1] (4)
method = "range"는 데이터를 [0, 1] 사이의 값으로 조정한다
un_iris_t = predict(un_iris, iris)
head(un_iris_t)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 0.22222222 0.6250000 0.06779661 0.04166667 setosa
## 2 0.16666667 0.4166667 0.06779661 0.04166667 setosa
## 3 0.11111111 0.5000000 0.05084746 0.04166667 setosa
## 4 0.08333333 0.4583333 0.08474576 0.04166667 setosa
## 5 0.19444444 0.6666667 0.06779661 0.04166667 setosa
## 6 0.30555556 0.7916667 0.11864407 0.12500000 setosa
Box-Cox transformation
Box-Cox 변환은 데이터가 한 쪽으로 쏠려있을 때 종모양에 가까운 형태로 데이터를 조정할 수 있게 해준다. 또는 비선형인 데이터를 선형으로 변환시키거나, 분산이 일정하지 않을 경우 사용할 수 있다.
bc_iris = preProcess(iris, method = "BoxCox")
bc_iris
## Created from 150 samples and 5 variables
##
## Pre-processing:
## - Box-Cox transformation (4)
## - ignored (1)
##
## Lambda estimates for Box-Cox transformation:
## -0.1, 0.3, 0.9, 0.6
lambda estimates를 보면 -0.1, 0.3, 0.9, 0.6 으로 되어있다. fudge 기본값 0.2로 인해 (1 - 0.2, 1 + 0.2) 구간은 1로 값이 변경되고 (0 - 0.2, 0 + 0.2)의 값은 0으로 값이 변경된다. lambda = 0일 경우에는 변환식의 분모가 0이기 때문에 해당 값의 자연로그를 취한다. 따라서 Lambda estimates가 -0.1인 Sepal.Length는 log(iris$Sepal.Length)와 값다. Labmda estimates가 0.9인 Petal.Length는 값이 변하지 않는다
predict 함수를 통해 실제로 변환시킨 값을 얻을 수 있다
bc_iris_t = predict(bc_iris, iris)
head(bc_iris_t)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 1.629241 1.520660 1.4 -1.0321154 setosa
## 2 1.589235 1.301297 1.4 -1.0321154 setosa
## 3 1.547563 1.391905 1.3 -1.0321154 setosa
## 4 1.526056 1.347113 1.5 -1.0321154 setosa
## 5 1.609438 1.561856 1.4 -1.0321154 setosa
## 6 1.686399 1.680826 1.7 -0.7048667 setosa
boxcox 변환에서는 fudge 옵션의 값을 변화시키면 tolerance 수준을 변경할 수 있다. 위에서 -0.1과 0.9가 있었기 때문에 두 변수 모두 값을 변화시킬 수 있도록 값을 0으로 둔다
bc_iris_nofudge = preProcess(iris, method = "BoxCox", fudge = 0)
bc_iris_nofudge
## Created from 150 samples and 5 variables
##
## Pre-processing:
## - Box-Cox transformation (4)
## - ignored (1)
##
## Lambda estimates for Box-Cox transformation:
## -0.1, 0.3, 0.9, 0.6
bc_iris_nofudge_t = predict(bc_iris_nofudge, iris)
head(bc_iris_nofudge_t)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 1.503443 1.520660 0.3929751 -1.0321154 setosa
## 2 1.469384 1.301297 0.3929751 -1.0321154 setosa
## 3 1.433760 1.391905 0.2959291 -1.0321154 setosa
## 4 1.415318 1.347113 0.4893297 -1.0321154 setosa
## 5 1.486601 1.561856 0.3929751 -1.0321154 setosa
## 6 1.551869 1.680826 0.6801608 -0.7048667 setosa
모든 수치형 변수의 값이 변화되었다
uspop 데이터에 Box-Cox Transformation 적용하기
우선 변수를 변환하기 전에 원본 데이터의 분포를 살펴보려고 한다.
plot(density(uspop))
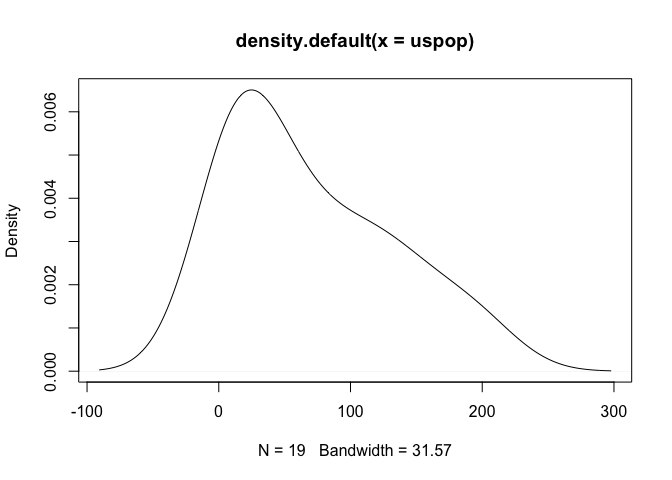
오른쪽 꼬리가 비교적 길다는 것을 확인할 수 있다. preProcess 함수를 적용하기 위해서 데이터를 data.frame 형태로 구성한다.
df_uspop = data.frame(
pop = as.numeric(uspop)
)
preProcess를 통해 box-cox 변환을 적용한다
bc_uspop = preProcess(df_uspop, method = "BoxCox")
bc_uspop
## Created from 19 samples and 1 variables
##
## Pre-processing:
## - Box-Cox transformation (1)
## - ignored (0)
##
## Lambda estimates for Box-Cox transformation:
## 0.2
predict를 통해 실제 데이터를 변환한다
bc_uspop_t = predict(bc_uspop, df_uspop)
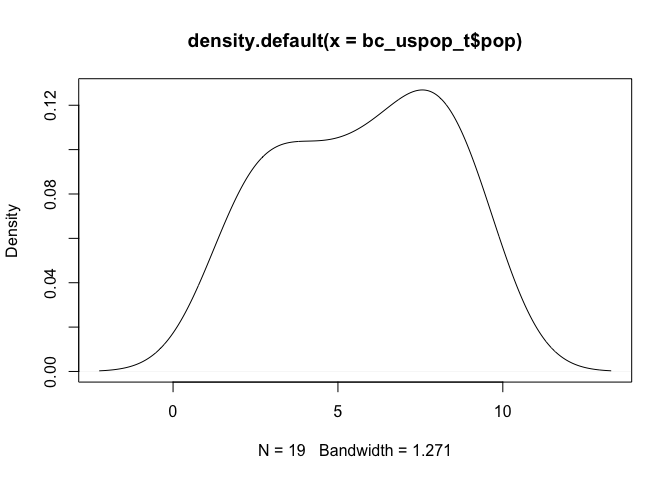
원래의 데이터와 비교하면 한 쪽에 데이터가 쏠리는 현상이 완화되었다.
plot(density(bc_uspop_t$pop))
다양한 전처리 방식 한번에 적용하기
preProcess 함수에서는 method 항목에 여러 요소를 한꺼번에 입력하면 여러 가지 전처리 방식을 한번에 적용할 수 있다.
multi_iris = preProcess(iris, method = c("center", "scale", "pca"))
# 사실 "pca"만 적용해도 center와 scale까지 한꺼번에 적용된다
multi_iris
## Created from 150 samples and 5 variables
##
## Pre-processing:
## - centered (4)
## - ignored (1)
## - principal component signal extraction (4)
## - scaled (4)
##
## PCA needed 2 components to capture 95 percent of the variance
PCA를 적용하고 나면 차원을 축소시켜 두 개의 변수를 남길 수 있다
multi_iris_t = predict(multi_iris, iris)
head(multi_iris_t)
## Species PC1 PC2
## 1 setosa -2.257141 -0.4784238
## 2 setosa -2.074013 0.6718827
## 3 setosa -2.356335 0.3407664
## 4 setosa -2.291707 0.5953999
## 5 setosa -2.381863 -0.6446757
## 6 setosa -2.068701 -1.4842053
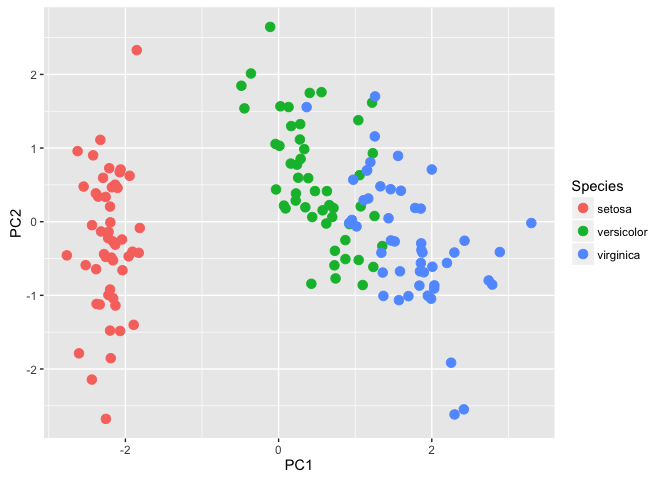
PCA를 적용시킨 iris데이터를 가지고 scatter plot을 그리면 다음과 같다
library(ggplot2)
ggplot(multi_iris_t, aes(x = PC1, y = PC2, colour = Species)) +
geom_point(size = 3)
Near Zero Variance
nearZeroVar는 값이 하나밖에 존재하지 않는 변수나 몇 개 안되는 요소만 반복되는 변수를 파악한다
df_nzv = data.frame(
v1 = c(rep(1,21), 2),
v2 = c(rep(1,11), rep(2, 8), rep(3, 3)),
v3 = rep(1, 22)
)
nearZeroVar함수를 사용하면 제거해야 할 열의 index를 반환해준다
nearZeroVar(df_nzv)
## [1] 1 3
어떤 기준을 통해서 제거할 열을 선택했는지 살펴보려면 saveMetrics = TRUE옵션을 추가한다
nearZeroVar(df_nzv, saveMetrics = TRUE)
## freqRatio percentUnique zeroVar nzv
## v1 21.000 9.090909 FALSE TRUE
## v2 1.375 13.636364 FALSE FALSE
## v3 0.000 4.545455 TRUE TRUE
nearZeroVar의 옵션 중에서 freqRatio는 제일 많이 등장하는 값의 개수와 두 번째로 많이 등장하는 값 개수의 비율을 의미한다
v1의 경우 21/1 = 21
v2의 경우 11/8 = 1.375
v3의 경우 값이 한 가지밖에 없기 때문에 0
percentUnique (uniqueCut에서 비교하는 값)은 전체 데이터 중에서 겹치지 않는(unique한) 값의 비율을 나타낸다
v1의 경우 1, 2이므로 2/22 * 100 = 9.0909
v2의 경우 1, 2, 3 이므로 3/22 = 13.6364
v3의 경우 1 밖에 없기 때문에 1/22 = 4.5454
기본적으로 freqCut은 19, uniqueCut은 10을 기준으로 near zero variance를 판단한다. 옵션을 통해 기준이 되는 값을 변경할 수 있다
nearZeroVar(df_nzv, uniqueCut = 5, saveMetrics = TRUE)
## freqRatio percentUnique zeroVar nzv
## v1 21.000 9.090909 FALSE FALSE
## v2 1.375 13.636364 FALSE FALSE
## v3 0.000 4.545455 TRUE TRUE
nearZeroVar(df_nzv, uniqueCut = 5)
## [1] 3
nearZeroVar(df_nzv, freqCut = 1)
## [1] 1 3
nearZeroVar(df_nzv, freqCut = 1, saveMetrics = TRUE)
## freqRatio percentUnique zeroVar nzv
## v1 21.000 9.090909 FALSE TRUE
## v2 1.375 13.636364 FALSE FALSE
## v3 0.000 4.545455 TRUE TRUE
method = "nzv"를 통해 preProcess 함수에서도 nearZeroVar를 적용할 수 있다
pp_nzv = preProcess(df_nzv, method = "nzv")
pp_nzv
## Created from 22 samples and 2 variables
##
## Pre-processing:
## - ignored (0)
## - removed (2)
pp_nzv_t = predict(pp_nzv, df_nzv)
head(pp_nzv_t)
## v2
## 1 1
## 2 1
## 3 1
## 4 1
## 5 1
## 6 1
v1, v3이 제거되었다
method = "zv"를 사용하면 zero variance(변수의 모든 값이 같은 경우)만 제거한다
pp_zv = preProcess(df_nzv, method = "zv")
pp_zv
## Created from 22 samples and 1 variables
##
## Pre-processing:
## - ignored (0)
## - removed (1)
pp_zv_t = predict(pp_zv, df_nzv)
head(pp_zv_t)
## v1 v2
## 1 1 1
## 2 1 1
## 3 1 1
## 4 1 1
## 5 1 1
## 6 1 1
v3가 제거되었다
Missing Values
결측치를 다루는 방법에는 여러 가지가 있다. 결측치가 존재하는 열이나 행을 제거하거나 결측치를 특정 값으로 바꾸는 등의 방법이 있다. caret의 preProcess 함수는 knn, bagging, median 등의 방법을 이용해서 결측치를 다른 값으로 채울 수 있다. 기능을 사용하기 위해서는 다른 패키지를 추가로 설치해야 할 수도 있다
실습을 위해서 iris데이터를 복사하고 [2, 2]의 값을 NA로 변경했다. NA로 변경한 값의 실제 값은 3.0이다
df_na_iris = iris
df_na_iris[2,2] = NA
우선 K-nearest Neighbor를 이용해서 결측치를 채울 수 있다. 추가로 패키지를 요구할 수 있는데 경고가 뜰 경우 필요한 패키지를 설치하고 다시 실행하면 된다.
#install.packages("RANN")
knnimp_na = preProcess(df_na_iris, method = "knnImpute")
knnimp_na
## Created from 149 samples and 5 variables
##
## Pre-processing:
## - centered (4)
## - ignored (1)
## - 5 nearest neighbor imputation (4)
## - scaled (4)
knnimp_na_t = predict(knnimp_na, df_na_iris[1:4])
head(knnimp_na_t)
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## 1 -0.8976739 1.01136747 -1.335752 -1.311052
## 2 -1.1392005 0.78269713 -1.335752 -1.311052
## 3 -1.3807271 0.32535645 -1.392399 -1.311052
## 4 -1.5014904 0.09668612 -1.279104 -1.311052
## 5 -1.0184372 1.24003781 -1.335752 -1.311052
## 6 -0.5353840 1.92604882 -1.165809 -1.048667
knnImpute를 적용하면 centering과 scaling이 같이 적용된다
bagImpute를 사용하면 Bagged Tree Model을 통해 결측치를 예측하여 채워넣는다. 마찬가지로 다른 패키지를 추가로 설치해야할 수 있다.
#install.packages("ipred")
bagimp_na = preProcess(df_na_iris, method = "bagImpute")
bagimp_na
## Created from 149 samples and 5 variables
##
## Pre-processing:
## - bagged tree imputation (4)
## - ignored (1)
bagimp_na_t = predict(bagimp_na, df_na_iris)
head(bagimp_na_t)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.500000 1.4 0.2 setosa
## 2 4.9 3.253777 1.4 0.2 setosa
## 3 4.7 3.200000 1.3 0.2 setosa
## 4 4.6 3.100000 1.5 0.2 setosa
## 5 5.0 3.600000 1.4 0.2 setosa
## 6 5.4 3.900000 1.7 0.4 setosa
median 값을 이용해서 결측치를 채워넣을 수도 있다. 이 경우 가장 간단하고 빠르지만 비교적 부정확할 수 있다.
medimp_na = preProcess(df_na_iris, method = "medianImpute")
medimp_na
## Created from 149 samples and 5 variables
##
## Pre-processing:
## - ignored (1)
## - median imputation (4)
medimp_na_t = predict(medimp_na, df_na_iris)
head(medimp_na_t)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosa
caret의 preProcess 함수에 대한 문서와 데이터 전처리에 대해 잘 정리된 블로그 링크를 첨부했다. 추가적으로 필요한 내용이 있다면 큰 도움이 될 것 같다.

